摘要
蛋白-蛋白之间的联系在细胞内功能和生物学进程中非常重要,突变可能导致疾病的富集。作者使用机器学习模型来精确的评价单点或者多点突变对蛋白-蛋白亲和力之间的影响。最后mmCSM-PPI的Pearson’s系数可以达到0.75 (RMSE = 1.64 kcal/mol),blind测试集验证的结果为0.7(RMSE=2.06kcal/mol)。
1. 介绍
介绍了我觉得有用的信息主要是介绍了几个亲和力数据库: ThermomutDB, ProTherm, PROXiMATE 和SKEMPI。
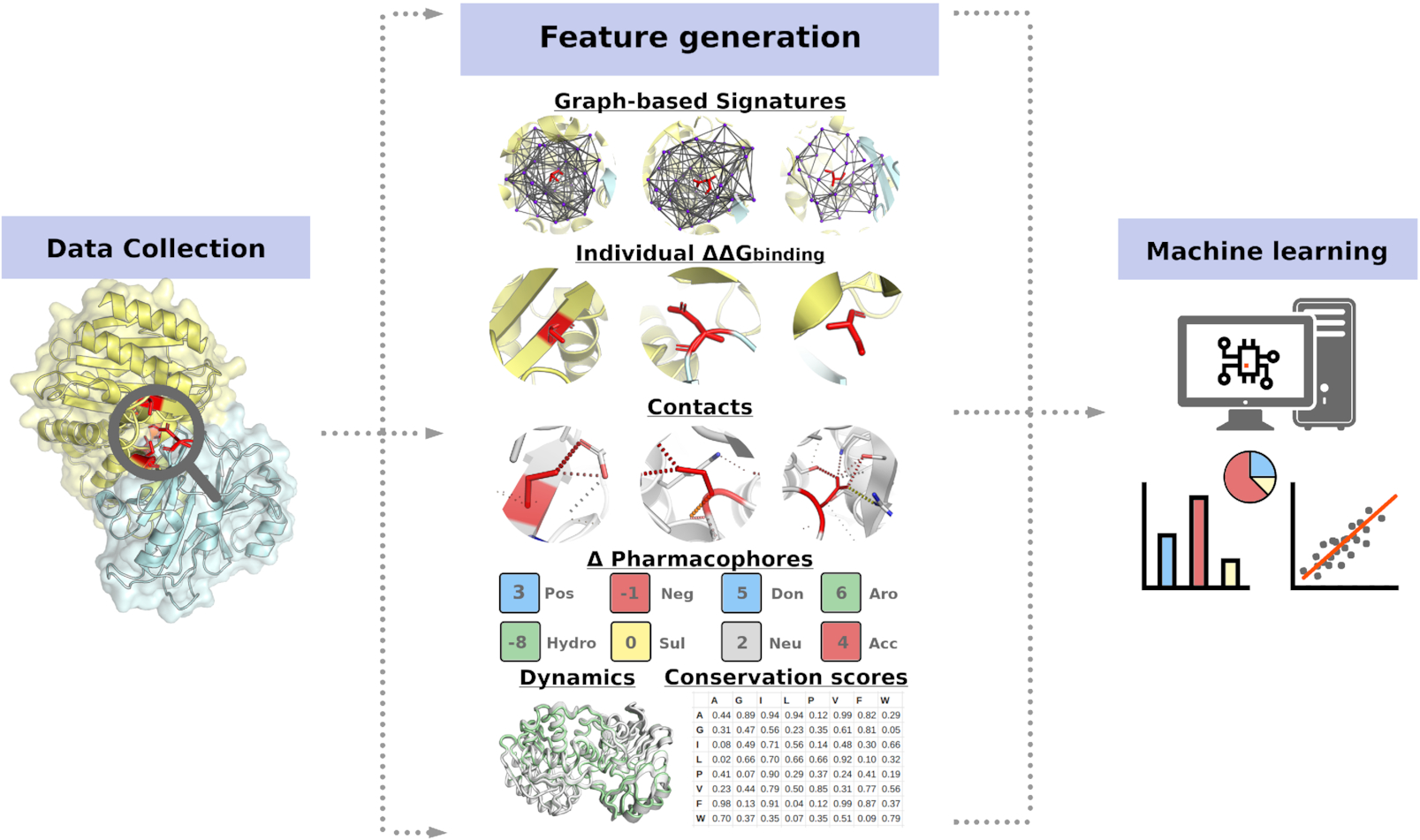
mmCSM-PPI的流程:其以SKEMPI2数据库为基础,通过其单点或者多点突变的结果和pdb数据来生成基于图签名的物理化学和几何参数,其在其中提取了6种特征参数(??):
- NMA获取的动力学属性
- 野生型残基环境
- 进化和接触式的打分
- 非共价联系
- 野生型的分子内距离
- 点突变的ΔΔGbinding
使用Scikit-learn 的GridSearch函数进行监督学习的算法优化
2. 材料和方法
2.1. 数据库
使用SKEMPI2
吉布斯自由能计算方法:
$$ \Delta G^{binding} = RTlin(K_{D})$$
R=1.9872 cak/K*mol, T为温度 KD为蛋白蛋白复合物亲和力
亲和力差值
$$ \Delta\Delta G^{binding}=\Delta G^{binding}WT- \Delta ^{binding} MT $$
2.2. 图基础的签名
?文章中没有讲清楚,待研究
2.3. 多突变建模
主要是6个主要的分类;
- 动力学,参考bio3d的normal mode analysis
- 残基环境, Biopython
- 保守性, AAindex
- 非共价联系, Arpeggio
- 分子内残基距离
- 预测的$$ \Delta \Delta G ^{binding} $$
2.4 机器学习
使用scikit-learn Python库进行cross-validation:
Extra Trees, Random Forest, Gradient Boosting, XGBoost
并使用Gridsearch函数来进行参数调优
3. 验证
其只和Discovery Studio, FoldX进行了比较,并未和Roestta ΔΔG 之类的进行比较,不过确实,Rosetta ΔΔG计算量过大。