Gromacs非标准残基构建
李老师其实已经有非常详细的实例来讲解如何进行非标准残基的搭建方法,此方法是之前根据李老师的方法结合amber教程进行改编的方法,可以作为参考。
我们使用ser磷酸化的蛋白作为实验,由于还未发表文章,暂时不提供示例步骤下载。若有问题可以邮件联系我交流:kangsgo#vip.qq.com
1.单独保存非标准残基
我们首先使用pymol将sep独立导出
1
2
|
select not resn SEP
remove sele
|
将其保存为sep.pdb文件,当然也可以直接用gedit等打开文件,将SEP部分(HETAM)copy导出
sep文件内容如下:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
|
ATOM 1 N SEP A 219 10.386 -7.407 38.687 1.00 31.68 N
ATOM 2 CA SEP A 219 10.979 -8.567 39.325 1.00 31.58 C
ATOM 3 C SEP A 219 12.450 -8.306 39.481 1.00 28.97 C
ATOM 4 O SEP A 219 13.189 -8.279 38.504 1.00 28.84 O
ATOM 5 CB SEP A 219 10.724 -9.808 38.495 1.00 0.00 C
ATOM 6 OG SEP A 219 11.474 -10.889 39.010 1.00 0.00 O
ATOM 7 P SEP A 219 11.419 -12.337 38.313 1.00 0.00 P1+
ATOM 8 O2P SEP A 219 12.339 -13.139 39.147 1.00 0.00 O1-
ATOM 9 O1P SEP A 219 9.990 -12.703 38.422 1.00 0.00 O1-
ATOM 10 O3P SEP A 219 11.893 -12.061 36.938 1.00 0.00 O1-
ATOM 11 H SEP A 219 10.767 -7.078 37.811 1.00 31.68 H
ATOM 12 HA SEP A 219 10.534 -8.738 40.305 1.00 31.58 H
ATOM 13 HB2 SEP A 219 9.662 -10.058 38.502 1.00 0.00 H
ATOM 14 HB3 SEP A 219 11.035 -9.614 37.467 1.00 0.00 H
|
不难看出带的电荷为-2
我们需要补全两端的氢键(封端),我是用gview补全的,也可以用pymol,chimera等补全,但是需要注意的是要查看补全的氢键是否正确,我在chimera上补全的氢键就有问题,对于补全氢键,个人的经验觉得正确度排序为:gv>chimera>pymol
补全后如下:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
|
HETATM 1 N SEP A 219 10.386 -7.407 38.687 N
HETATM 2 CA SEP A 219 10.979 -8.567 39.325 C
HETATM 3 C SEP A 219 12.450 -8.306 39.481 C
HETATM 4 O SEP A 219 13.189 -8.279 38.504 O
HETATM 5 CB SEP A 219 10.724 -9.808 38.495 C
HETATM 6 OG SEP A 219 11.474 -10.889 39.010 O
HETATM 7 P SEP A 219 11.419 -12.337 38.313 P
HETATM 8 O2P SEP A 219 12.339 -13.139 39.147 O
HETATM 9 O1P SEP A 219 9.990 -12.703 38.422 O
HETATM 10 O3P SEP A 219 11.893 -12.061 36.938 O
HETATM 11 H SEP A 219 10.767 -7.078 37.811 H
HETATM 12 HA SEP A 219 10.534 -8.738 40.305 H
HETATM 13 HB2 SEP A 219 9.662 -10.058 38.502 H
HETATM 14 HB3 SEP A 219 11.035 -9.614 37.467 H
HETATM 15 2H SEP A 219 10.544 -6.657 39.329 H
HETATM 16 H SEP A 219 12.857 -8.142 40.457 H
TER 16 SEP A 219
END
CONECT 1 2 11 15
CONECT 2 1 3 5 12
CONECT 3 2 4 16
CONECT 4 3
CONECT 5 2 6 13 14
CONECT 6 5 7
CONECT 7 6 8 9 10
CONECT 8 7
CONECT 9 7
CONECT 10 7
CONECT 11 1
CONECT 12 2
CONECT 13 5
CONECT 14 5
CONECT 15 1
CONECT 16 3
|
2.制作高斯输入文件
1
|
antechamber -fi pdb -i sep.pdb -fo gcrt -o sep.gjf -ch "sep.chk" -gm "%mem=2048MB" -gn "%nproc=4" -nc -2
|
如果按照李老师的方法还需要使用-ge 生成高斯esp文件,通过iop(6/50=1),可以参考下面一条命令
1
|
antechamber -fi pdb -i sep.pdb -o ligand.gjf -fo gcrt -pf y -gn "%nproc=8" -gm "%mem=1000MB" -ch "ligand" -gk "#HF/6-31G* SCF=tight Test Pop=MK iop(6/33=2) iop(6/42=6) iop(6/50=1) opt" -ge ligand.gesp -gv 1
|
命令解释如下:
-fi为输入格式
-i为输入文件
-fo为输出文件格式 gcrt为高斯gjf格式
-ch为高斯chk文件
-gm -gn为使用内存和核数
-nc 为电荷数
3.转化为gromacs文件
实际上与蛋白配体中的小分子制作方式是一样的,首先在这里下载ACPYPE,注意需要下载sf版本,否则而面角的表达方式不对,首先生成mol2文件
1
|
antechamber -fi gout -i sep.log -rn SEP -fo mol2 -o sep.mol2 -c resp -pf y -at amber
|
在这里最好修改一下原子名称,改成与sep.pdb对应,且需要对其进行修改,因为gromacs加氢从1开始,故将HB2,HB3改为HB1,HB2
最后的结果如下:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
|
...
@<TRIPOS>ATOM
1 N 3.0030 -1.1500 0.1620 N 1 SEP -1.141452
2 CA 1.8370 -0.4940 -0.4350 CT 1 SEP 0.462377
3 C 1.8020 0.9980 -0.1460 C 1 SEP 0.457721
4 O 2.8040 1.5940 0.1700 O 1 SEP -0.633121
5 CB 0.5190 -1.1870 -0.0550 CT 1 SEP 0.192426
6 OG -0.5100 -0.6800 -0.7990 OS 1 SEP -0.641051
7 P -1.7850 0.1390 0.0880 P 1 SEP 1.339193
8 O2P -2.7260 0.5020 -1.0190 O2 1 SEP -0.920441
9 O1P -2.2370 -0.9110 1.0650 O2 1 SEP -0.920441
10 O3P -1.0110 1.2850 0.7110 O2 1 SEP -0.920441
11 H 2.7710 -1.3680 1.1160 H 1 SEP 0.367588
12 HA 1.9150 -0.5690 -1.5210 H1 1 SEP -0.041482
13 HB1 0.6570 -2.2570 -0.2470 H1 1 SEP -0.007243
14 HB2 0.3490 -1.0790 1.0140 H1 1 SEP -0.007243
15 H2 3.7290 -0.4600 0.2230 H 1 SEP 0.367588
16 H1 0.8340 1.4820 -0.2400 HA 1 SEP 0.046022
....
|
生成的mol2文件使用parmchk2进行补全参数
1
|
parmchk2 -i sep.mol2 -f mol2 -o sep.frcmod
|
创建leap.in文件,输入如下内容(注意sep修改成自己的名称):
1
2
3
4
5
6
7
|
source leaprc.ff99SBildn
source leaprc.gaff
loadamberparams sep.frcmod
lig=loadmol2 sep.mol2
check lig
saveamberparm lig sep.prmtop sep.inpcrd
quit
|
运行
运行acpype
1
|
python2.7 acpype.py -p sep.prmtop -x sep.inpcrd -d
|
将会生成SEP_GMX.top和SEP_GMX.gro两个文件,其中SEP_GMX.gro我们用不着
4.整理残基的rtp条目
为了保存力场rtp信息,我们需要创建一个sep.rtp文件
我们需要对SEP_GMX.gro文件进行整理,即把虚拟的原子电荷附加到N端头和C端尾部的位置。
如:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
|
[ atoms ]
; nr type resi res atom cgnr charge mass ; qtot bond_type
1 N 1 SEP N 1 -1.141453 14.01000 ; qtot -1.141
2 CT 1 SEP CA 2 0.462377 12.01000 ; qtot -0.679
3 C 1 SEP C 3 0.457721 12.01000 ; qtot -0.221
4 O 1 SEP O 4 -0.633122 16.00000 ; qtot -0.854
5 CT 1 SEP CB 5 0.192426 12.01000 ; qtot -0.662
6 OS 1 SEP OG 6 -0.641052 16.00000 ; qtot -1.303
7 P 1 SEP P 7 1.339196 30.97000 ; qtot 0.036
8 O2 1 SEP O2P 8 -0.920442 16.00000 ; qtot -0.884
9 O2 1 SEP O1P 9 -0.920442 16.00000 ; qtot -1.805
10 O2 1 SEP O3P 10 -0.920442 16.00000 ; qtot -2.725
11 H 1 SEP H 11 0.367588 1.00800 ; qtot -2.358
12 H1 1 SEP HA 12 -0.041482 1.00800 ; qtot -2.399
13 H1 1 SEP HB1 13 -0.007243 1.00800 ; qtot -2.406
14 H1 1 SEP HB2 14 -0.007243 1.00800 ; qtot -2.414
15 H 1 SEP H2 15 0.367588 1.00800 ; qtot -2.046
16 HA 1 SEP H1 16 0.046022 1.00800 ; qtot -2.000
|
1 N的电荷应该变为-1.141453+0.367588
3 C的电荷应该变为0.457721+0.046022
然后用我写的脚本进行内容的提取:
首先创建config.txt文件,在其中写入需要删除的虚拟原子(每一行一个)
然后运行
1
|
python rtp.py SEP_GMX.top SEP>data.txt
|
结果如下:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
|
[ bondedtypes ]
; bonds angles dihedrals impropers all_dihedrals nrexcl HH14 RemoveDih
1 1 9 4 1 3 1 0
[ SEP ]
[ atoms ]
N N -0.773865 1
...
[ bonds ]
N CA 1.4620e-01 2.7506e+05
N H 1.0130e-01 3.3740e+05
CA C 1.5240e-01 2.6192e+05
...
[ angles ]
N CA C 1.0906e+02 5.6066e+02
N CA CB 1.1161e+02 5.5153e+02
N CA HA 1.0888e+02 4.1706e+02
CA N H 1.1768e+02 3.8325e+02
CA C O 1.2320e+02 5.6400e+02
...
[ dihedrals ] ; propers
N CA C O 180.00 0.00000 9
N CA CB OG 0.00 0.65084 9
N CA CB HB1 0.00 0.65084 9
N CA CB HB2 0.00 0.65084 9
...
[ impropers ]
|
我们需要在[bonds]中增加前一个氨基酸残基的联系连接,[impropers]中增加前后氨基酸残基的联系连接,可以简单的看非标准残基来源的氨基酸残基模板(aminoacids.rtp)或者查看SEP_GMX.top文件中删除的两个氢原子的部分,最后整理结果如下:
1
2
3
4
5
6
7
8
9
10
11
|
...
[ bonds ]
-C N
N CA 1.4620e-01 2.7506e+05
N H 1.0130e-01 3.3740e+05
CA C 1.5240e-01 2.6192e+05
CA CB 1.5380e-01 2.5179e+05
...
[ impropers ]
-C CA N H
CA +N C O
|
5.整理残基的hdb条目
由于氢键一般有问题,我们需要重新绘制氢键,那么我们创建一个sep.hdb的文件,按照下图来进行设置(图片来源李老师博客):
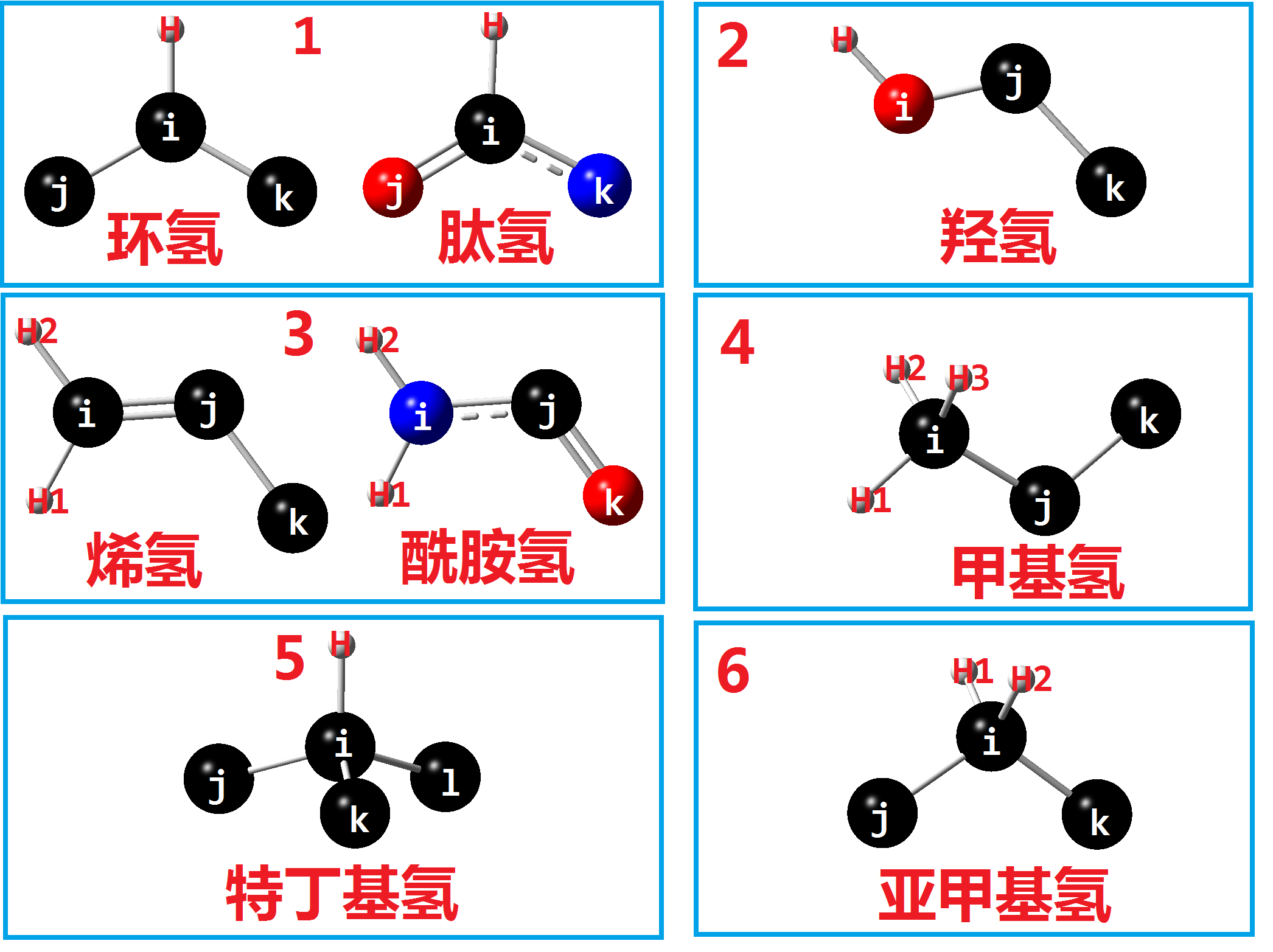
结果如下图(可以抄SER的信息):
1
2
3
4
|
SEP 3
1 1 H N -C CA
1 5 HA CA N CB C
2 6 HB CB CA OG
|
6.模拟尝试
将gromacs 目录下(gromacs/share/gromacs/top)中的amber99sb-ildn.ff拷贝至实验目录下(如果找不到可以用which gmx_mpi),将sep.rtp文件和sep.hdb文件放到拷贝出来的amber99sb-ildn.ff目录下。且在top目录下的residuetypes.dat中申明SEP为蛋白,即:
1
2
3
4
5
6
7
8
9
10
|
SEP Protein ; sep,增加部分
ABU Protein
ACE Protein
AIB Protein
ALA Protein
ARG Protein
ARGN Protein
ASN Protein
ASN1 Protein
...
|
最后进行模拟尝试看是否可以跑通
1
|
gmx_mpi pdb2gmx -f model.pdb -o model_process.gro -water tip3p -ignh
|
若提示前一个残基或者末端残基报错,那么检查是否是上述两个文件出错,若提示缺失(SEP的)原子,那么创建sep.atp文件,在SEP_GMX.top,找到提示的缺失原子,放入该文件中,最后将sep.atp文件放到拷贝出来的amber99sb-ildn.ff目录下。
参考资料:
GROMACS非标准残基教程2-芋螺毒素小肽实例
向gromacs中添加小分子力场方法
amber中非标准氨基酸残基的参数生成
Simulating the Green Fluorescent Protein
使用AmberTools+ACPYPE+Gaussian创建小分子GAFF力场的拓扑文件
